-
Schuster, Stefan, Univ.-Prof. Dr. Professur für Bioinformatik
Raum 3403
Ernst-Abbe-Platz 1-2
07743 Jena
-
Dimitriew, Wassili Professur für Bioinformatik
Raum 3427
Ernst-Abbe-Platz 1-2
07743 Jena
-
Projektbeschreibung
Aufgrund der Evolution sind die Charakteristika wie Stoffwechsel und Zellverhalten eines Organismus hochgradig optimiert. In diesen Projekt nutzen wir die dynamische Optimierung, welche ursprünglich im Ingenieurwesen angewendet wurde, um zeitabhängige Optimalitätsprinzipien von biologischen Systemen zu verstehen. Im Speziellen wollen wir die Prinzipien sowohl bei der Regulation des Zellstoffwechsels, als auch bei der Interaktion von Krankheitserregern und dem Immunsystem entschlüsseln. Da dieses Projekt eingebunden ist in den Transregio “FungiNet”, sichert die enge Kooperation mit Experimentatoren die Validierung der neugewonnenen Hypothesen.
-
Ausgewählte Literatur
- W. Dimitriew, S. Schuster
Dynamic optimization elucidates higher-level pathogenicity strategies of Pseudomonas aeruginosa
µLife, 2025, accepted - J. Ewald, F. Rivieccio, L. Radosa, S. Schuster, A.A. Brakhage, C. Kaleta
Dynamic optimization reveals alveolar epithelial cells as key mediators of host defense in invasive aspergillosis
PLOS Comp. Biol. 17, 2021, e1009645 - Ewald, M. Bartl, C. Kaleta
Deciphering the regulation of metabolism with dynamic optimization: an overview of recent advances
Biochemical Society Transactions 45 (4), 2017, 1-9 - Ewald, M. Bartl, T. Dandekar, C. Kaleta
Optimality principles reveal a complex interplay of intermediate toxicity and kinetic efficiency in the regulation of prokaryotic metabolism
PLOS Computational Biology 13, 2017, e100537 - Jan Ewald, Martin Kötzing, Martin Bartl, Christoph Kaleta
Footprints of Optimal Protein Assembly Strategies in the Operonic Structure of Prokaryotes
Metabolites 2015, 5(2), 252-269; doi:10.3390/metabo5020252 - F. Wessely, M. Bartl, R. Guthke, P. Li, S. Schuster, C. Kaleta
Optimal regulatory strategies for metabolic pathways in Escherichia coli depending on protein costs.
Molecular Systems Biology 7, 2011, 515
- W. Dimitriew, S. Schuster
-
Finanzierung
DFG Collaborative Research Center / Transregio 124 "FungiNet"
-
Projektbeschreibung
Ein Schlüssel zur Pathogenität von Mikroorganismen ist ihr flexibler und facettenreicher Metabolismus, der es ihnen ermöglicht, im Wirt zu überleben und zu wachsen. In diesem Projekt suchen wir mit verschiedenen mathematischen und informatischen Methoden, mögliche Schwachpunkte im Metabolismus von Pathogenen. Insbesondere nutzen wir toxische Zwischenprodukte von Stoffwechselwegen in pathogenen Pilzen wie Candida albicans, um Angriffspunkte für neue Antimykotika zu finden.
-
Ausgewählte Literatur
- J. Ewald, P. Sieber, R. Garde, S.N. Lang, S. Schuster, B. Ibrahim
Trends in mathematical modeling of host-pathogen interactions
Cell. Mol. Life Sci. Volume 77, Issue 3, 2020, pp 467–480 - Ewald, M. Bartl, T. Dandekar, C. Kaleta
Optimality principles reveal a complex interplay of intermediate toxicity and kinetic efficiency in the regulation of prokaryotic metabolism
PLOS Computational Biology 13, 2017, e100537 - S. Dühring, S. Germerodt, C. Skerka, P. F. Zipfel, T. Dandekar, S. Schuster
Host-pathogen interactions between the human innate immune system and Candida albicans - Understanding and modeling defense and evasion strategies
Frontiers in Microbiology 6 (625), 2015
- J. Ewald, P. Sieber, R. Garde, S.N. Lang, S. Schuster, B. Ibrahim
-
Finanzierung
DFG Collaborative Research Center / Transregio 124 "FungiNet"
-
Projektbeschreibung
Eine Besonderheit von schnellwachsenden Zellen, wie bspw. Krebszellen, ist die unvollständige Verstoffwechselung von Glykose zu Laktat, in Krebszellen Warburg-Effekt genannt. Obwohl die Energieausbeute pro Molekül geringer ist als die vollständige Veratmung zu CO2, kann man mit linearen Optimierungsmodellen zeigen, dass dieses Verhalten optimal ist in Hinblick der Enzymkosten und Energierate. In erweiterten Modellen untersuchen wir weitere Energiequellen wie Glutamin, um die zusätzliche Aufnahme und Verstoffwechselung anderer Kohlenstoffquellen von Krebszellen zu verstehen.
-
Ausgewählte Literatur
- J. Ewald, Z. He, W. Dimitriew, S. Schuster
Including glutamine in a resource allocation model of energy metabolism in cancer and yeast cells.
npj Systems Biology and Applications 10, 77 (2024). - P. Möller, X. Liu, S. Schuster, D. Boley
Linear programming model can explain respiration of fermentation products.
PloS one 13, 2018, e0191803 - S. Schuster, D. Boley, P. Möller, H. Stark, C. Kaleta
Mathematical models for explaining the Warburg effect: A review focussed on ATP and biomass production
Biochemical Society Transactions 43 (6), 2015, 1187-1194 - S. Schuster, L. de Figueiredo, A. Schroeter, C. Kaleta
Combining Metabolic Pathway Analysis with Evolutionary Game Theory. Explaining the occurrence of low-yield pathways by an analytic optimization approach
BioSystems 105, 2011, 147-153
- J. Ewald, Z. He, W. Dimitriew, S. Schuster
-
Projektbeschreibung
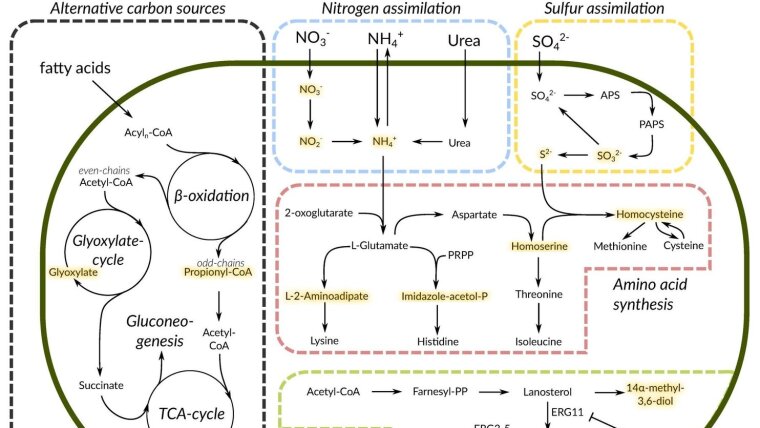
Stoffwechselmodelle im Genommaßstab sind nützlich, da sie Aufschluss darüber geben können, wie die Vielzahl von Reaktionen in lebenden Zellen miteinander verbunden ist und wie sich Änderungen einiger Reaktionen auf das gesamte Netzwerk auswirken können. Wir konzentrieren uns auf die Erstellung eines genomweiten Stoffwechselmodells des pathogenen Pilzes Lichtheimia corymbifera. Laut WHO gehört dieser Pilz zu einer Gruppe humanpathogener Krankheitserreger mit hoher Priorität und kann schwere Krankheiten verursachen. Gleichzeitig wird der Pilz in vielen Kulturen aktiv zur Fermentierung verwendet, ohne dass offensichtliche Nebenwirkungen auftreten. Unser Ziel ist es, diese Diskrepanz anhand der verfügbaren Daten zum Stoffwechsel von L. corymbifera zu erklären.
In dieser Arbeit verwenden wir Toolboxen für Modellrekonstruktionen im Genommaßstab, stoffwechselbezogene Datenbanken und Pakete für die Interaktion mit ihnen sowie große Sprachmodelle, um Tausende von Reaktionen einzuordnen, die im lebenden Organismus auftreten.
-
Ausgewählte Literatur
Wird später hinzugefügt