-
Schuster, Stefan, Univ.-Prof. Dr. Professur für Bioinformatik
Raum 3403
Ernst-Abbe-Platz 1-2
07743 Jena
-
Wesp, Valentin Professur für Bioinformatik
Raum 3422A
Ernst-Abbe-Platz 1-2
07743 Jena
-
Malycheva, Tatjana Professur für Bioinformatik
Raum 3429
Ernst-Abbe-Platz 1-2
07743 Jena
-
Lovate, Gabriel Lencioni Professur für Bioinformatik
Raum Raum 3402; 4. Etage
Ernst-Abbe-Platz 2
07743 Jena
-
Projektbeschreibung
Der Schwerpunkt des Projekts liegt auf altersbedingten Proteinschäden, die zu Aggregaten führen. Diese können nicht mehr abgebaut werden und reichern sich an, bis die Zelle funktionell stark eingeschränkt ist und schließlich die Apoptose einleitet. Ziel ist es, die am meisten oder auch am wenigsten anfälligen Ziele in Bezug auf oxidativen Schaden zu ermitteln. Auch strukturelle Informationen und bekannte Aggregatproteine werden einbezogen. Weiterhin werden funktionelle Zusammenhänge und Domänen in diesem Kontext untersucht.
-
Ausgewählte Literatur
- M. Fichtner, S. Schuster, H. Stark
Influence of spatial structure on protein damage susceptibility: a bioinformatics approach
Sci. Rep. 11, 2021, 4938 - E. Barth, P. Sieber, H. Stark, S. Schuster
Robustness during Aging — Molecular Biological and Physiological Aspects
Cells 9 (8), 2020, 1862 - M. Fichtner, S. Schuster, H. Stark
Determination of scoring functions for protein damage susceptibility
BioSystems 187, 2020, 104035, epub ahead of print - S. Schäuble, K. Klement, S. Marthandan, S. Münch, I. Heiland, S. Schuster, P. Hemmerich, S. Diekmann
Quantitative model of cell cycle arrest and cellular senescence in primary human fibroblasts.
PLoS ONE 7, 2012, e42150
- M. Fichtner, S. Schuster, H. Stark
-
Projekbeschreibung
Antibiotikaresistenzgene (ARGs) und biosynthetische Gencluster (BGCs) spielen eine entscheidende Rolle in mikrobiellen Gemeinschaften, indem sie die Produktion antimikrobieller Substanzen und die Anpassung an selektive Umweltbedingungen beeinflussen. In diesem Projekt untersuchen wir, wie Umweltveränderungen, mikrobieller Wettbewerb und genetische Mobilität die Koevolution von ARGs und BGCs über verschiedene Ökosysteme hinweg antreiben. Wir analysieren metagenomische Datensätze aus unberührten sowie anthropogen beeinflussten Lebensräumen mithilfe einer maßgeschneiderten Nextflow-Pipeline, die Werkzeuge zur Erkennung von BGCs (antiSMASH) mit Frameworks zur ARG-Erkennung (AMRFinderPlus, MEGARes, ResFinder) integriert. Nach Validierung werden wir die Analyse auf weitere Datensätze ausweiten, um die Verbreitung genomischer Merkmale unter unterschiedlichen Umweltbedingungen zu bewerten. Der Fokus liegt zunächst auf Metagenomik, soll aber zukünftig um metatranskriptomische und metabolomische Ansätze erweitert werden. Unsere Ergebnisse könnten Strategien zur verantwortungsvollen Nutzung von Antibiotika verbessern und die Bedeutung unberührter Lebensräume als Reservoirs für neuartige antimikrobielle Substanzen hervorheben.
-
Ausgewählte Literatur
- T.C. Borelli, G.L. Lovate, A.F.T. Scaranello, L.F. Ribeiro, L. Zaramela,F. M. Pereira-dos-Santos, R. Silva-Rocha, M.E. Guazzaroni
Combining Functional Genomics and Whole-Genome Sequencing to Detect Antibiotic Resistance Genes in Bacterial Strains Co-Occurring Simultaneously in a Brazilian HospitalExterner Link
Antibiotics 10 (4), 419 - L.F. Alves, C.A. Westmann, G.L. Lovate, G.M.V. de Siqueira, T.C. Borelli, M.E. Guazzaroni
Metagenomic approaches for understanding new concepts in microbial scienceExterner Link
International journal of genomics 2018 (1), 2312987
- T.C. Borelli, G.L. Lovate, A.F.T. Scaranello, L.F. Ribeiro, L. Zaramela,F. M. Pereira-dos-Santos, R. Silva-Rocha, M.E. Guazzaroni
-
Projektbeschreibung
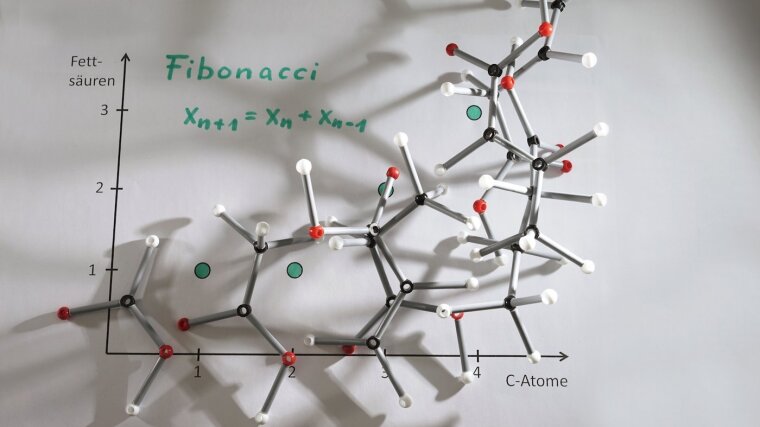
In diesem Projekt werden zum einen die kombinatorischen Möglichkeiten aliphatischer Aminosäuren und Fettsäuren untersucht. Hierzu werden Molekülklassen mit bestimmten Einschränkungen gewählt, da eine erschöpfende Auflistung nicht praktikabel ist. Es werden mathematische Berechnungen wie z.B. Rekursionen zu den erhaltenen Zahlenfolgen durchgeführt. Z.B. ergibt sich bei der Frage, wie viele unverzweigte, unmodifizierte Fettsäuren mit n C-Atomen es prinzipiell geben kann, die berühmte Fibonacci-Folge. Bei modifizierte Fettsäuren sind es verallgemeinerte Fibonacci-Zahlen. Zusätzlich wird durch Literaturrecherche verifiziert, welche Kombinationen real in Organismen synthetisiert werden. Zum anderen werden unterschiedliche Klassifikationen von Aminosäuren entwickelt, die bestehende Verfahren ergänzen oder optimieren sollen. Dadurch erhoffen wir uns, Einsichten zu grundlegenden Fragen der biochemischen Evolution erhalten.
-
Ausgewählte Literatur
- S. Schuster, T. Malycheva
Enumeration of saturated and unsaturated substituted N-heterocycles
arXiv:2309.02343, 2023, Preprint - A. Then, H. Zhang, B. Ibrahim, S. Schuster
Bioinformatics analysis of the periodicity in proteins with coiled-coil structure—Enumerating all decompositions of sequence periods
Intern. J. Molec. Sci. 23, 2022, 8692 - A. Then, K. Mácha, B. Ibrahim, S. Schuster
A novel method for achieving an optimal classification of the proteinogenic amino acids
Scientific Reports 10, 2020, 15321
https://www.nature.com/articles/s41598-020-72174-5Externer Link - M. Fichtner, K. Voigt, S. Schuster
The tip and hidden part of the iceberg: Proteinogenic and non-proteinogenic aliphatic amino acids
Biochimica and Biophysica Acta - General Subjects 1861 (1, Part A), 2017, 3258-3269
https://www.ncbi.nlm.nih.gov/pubmed/27554846Externer Link - S. Schuster, M. Fichtner, S. Sasso
Use of Fibonacci numbers in lipidomics - Enumerating various classes of fatty acids
Scientific Reports 7, 2017, 39821
https://www.ncbi.nlm.nih.gov/pubmed/28071669Externer Link - K. Grützmann, S. Böcker, S. Schuster
Combinatorics of aliphatic amino acids
Naturwissenschaften 98, 2011, 79-86
https://link.springer.com/article/10.1007/s00114-010-0743-2Externer Link
- S. Schuster, T. Malycheva
-
Projektbeschreibung
Hoch entwickelte Eukaryoten aber auch Pilze können mRNA in unterschiedlicher Weise spleißen. Doch es ist nicht bekannt, in wie weit dieser Mechanismus bei Pilzen verbreitet ist und welche zellulären Prozesse davon betroffen sind. Spielt alternatives Spleißen eine Rolle bei der Unterscheidung zu einzelligen Pilzen oder beeinflusst es die Pathogenität?
Wir analysieren RNA-Seq-Daten, um Antworten auf diese Fragen zu finden. Dabei sind vor allem Pilzspezies wie Candida albicans, Aspergillus fumigatus und Cryptococcus neoformans für uns von großem Interesse. Wir verwenden verschiedene Software, um alternativ gespleißte Gene zu bestimmen. Diese werden anschließend näher betrachtet, um Rückschlüsse auf die biologische Funktion des alternativen Spleißens zu ziehen. Dabei wird vor allem auch die Regulierung in verschiedenen Pilzspezies betrachtet und der phylogenetische Zusammenhang analysiert. -
Ausgewählte Literatur
- P. Sieber, M. Platzer, S. Schuster
The definition of open reading frame revisited
Trends in Genetics 34, 2018, 167-170 - V. Wesp, G. Theißen, S. Schuster
Statistical analysis of synonymous and stop codons in pseudo-random and real sequences as a function of GC content
Sci. Rep. 13, December, 2023, 22996 - P. Sieber*, E. Barth*, M. Marz
The landscape of the alternatively spliced transcriptome remains stable during aging across different species and tissues
bioRxiv February, 2019, 541417 - P. Sieber, K. Voigt, P. Kämmer, S. Brunke, S. Schuster, J. Linde
Comparative study on alternative splicing in human fungal pathogens suggests its involvement during host invasion
Frontiers in Microbiology 9, 2018, 2313 - P. Sieber, M. Platzer, S. Schuster
The definition of open reading frame revisited.
Trends in Genetics 34, 2018, 167-170 - J. Linde, S. Duggan, M. Weber, F. Horn, P. Sieber, D. Hellwig, K. Riege, M. Marz, R. Martin, R. Guthke, O. Kurzai
Defining the transcriptomic landscape of Candida glabrata by RNA-Seq.
Nucleic acids research 43, 2015, 1392-1406
- P. Sieber, M. Platzer, S. Schuster