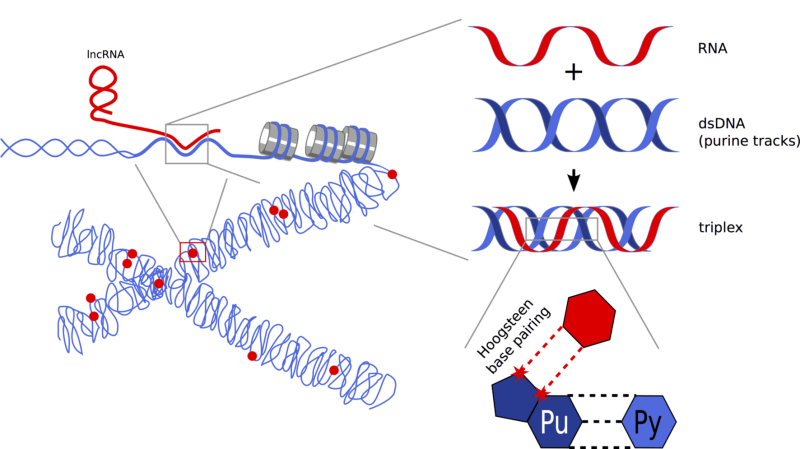
Genom-weite Studie haben gezeigt, dass Proteine-kodieren Sequenzen nur einen sehr geringe Anteil an eukaryotischen Genomen ausmachen (im Menschen etwa 1.5%). Dennoch wird nahezu die gesamte DNA transkribiert was zu tausenden von nicht-kodierenden-RNAs (ncRNA) führt. Im Laufe des letzten Jahrzehnts wurde die biologische Relevanz dieser ncRNAs intensiv untersucht, und es wurden verschiedene regulatorische Funktionen im Zellkern und Cytoplasma nachgewiesen. Eine der interessantesten Eigenschaften von ncRNAs ist die Fähigkeit, die Chromatinstruktur zu beeinflussen. In den meisten Fällen ist der zugrundeliegende Mechanismus, gerade im Hinblick auf die Erkennung der Zielregionen im Genom, noch unverstanden.
Ziel unserer Forschungsgruppe ist es, ncRNA:DNA-Triplexe zu identifizieren, detailliert zu beschreiben und ihre Rolle beim Aufbau der Chromatinlandschaft zu ergründen. In einer Triplexstruktur bindet die ncRNA in die große Furche der DNA-Doppelhelix durch die Ausbildung von Hoogsteen-Wasserstoffbrückenbindungen. Daraus ergibt sich eine Sequenz-Spezifität, so dass Triplex-formierende ncRNAs in cis oder trans an einem oder mehreren Genen fungieren können. Diese Eigenschaften, zusammen mit der nicht invasiven Interaktion mit der DNA, machen den ncRNA:DNA-Triplex zu einem attraktiven Mechanismus mit dem ncRNAs ihre Zielgene direkt finden können.
Unser Labor erforscht, wie Triplexe sich gegen Proteinbindung oder gegen die Bildung alternativer Nukleinsäurestrukturen an den Zielorten behaupten können, und wie/welche Chromatin-assoziierten Faktoren mit den Triplexstrukturen zusammenwirken. Mit Untersuchungen an Modell-Genen sowie mit genomweiten Hochdurchsatzstudien hoffen wir, ein grundlegendes Verständnis der ncRNA-vermittelten Triplex-Bildung zu erzielen. Vor dem Hintergrund der großen Anzahl an genetischen Loci, die das Potenzial haben Triplexe zu bilden, sind wir überzeugt, dass ncRNA:DNA-Triplexe wichtige und im Zellkern weit verbreitete Strukturen sind, die epigenetische Regulation vermitteln.
Triplex
Foto: Holger Bierhoff